CryoSPARC是当前冷冻电镜单颗粒重构的主流软件之一,它的界面交互十分友好,能够快速上手,且处理的速度和质量也非常高。本文采用一套铁蛋白apoferritin的数据(empiar-10200)从中选择了26张照片,我们服务器在/data/rawdata/tutorial/apoferritin下。
新建
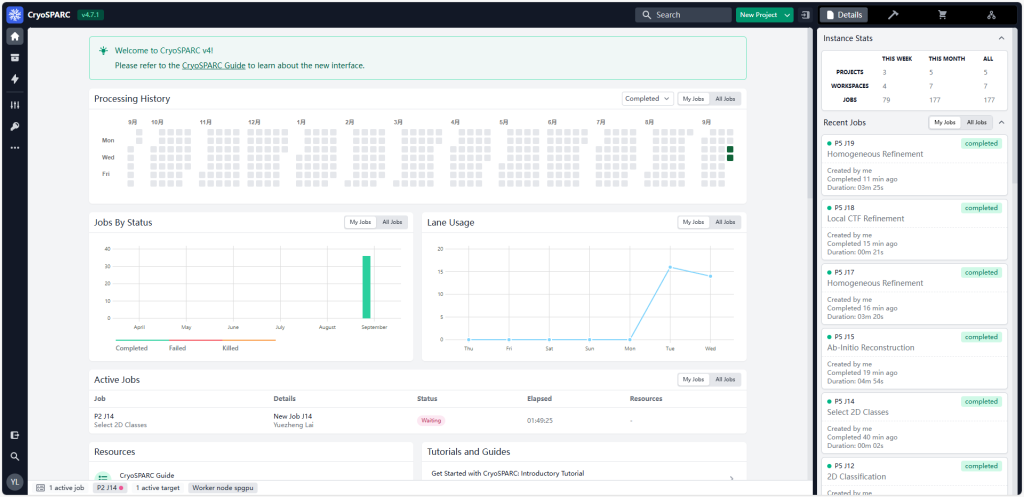
这是CryoSPARC v4.7.1的主界面,CryoSPARC用户端通过网页访问,网址一般是服务器的ip地址加上端口号(默认是39000)。
点击上方绿色的New Project按钮可以新建Project。
Title输入你想取的名称,然后选择Project保存的路径(在数据盘里创建一个自己的文件夹),它会在选择的文件夹内创建一个”CS-Title”的文件夹,所有的运算结果都在这个文件夹下。点击Create新建。
进入Project之后,创建一个Workspace,为某套数据取一个名字。不同worksparce之间是可以通过link job来互相访问计算的结果的,所以自己的类似蛋白的多套数据最好放在一个Project里,方便引用模板匹配或者三维模型,分成不同Workspace方便分类和查找自己的任务。
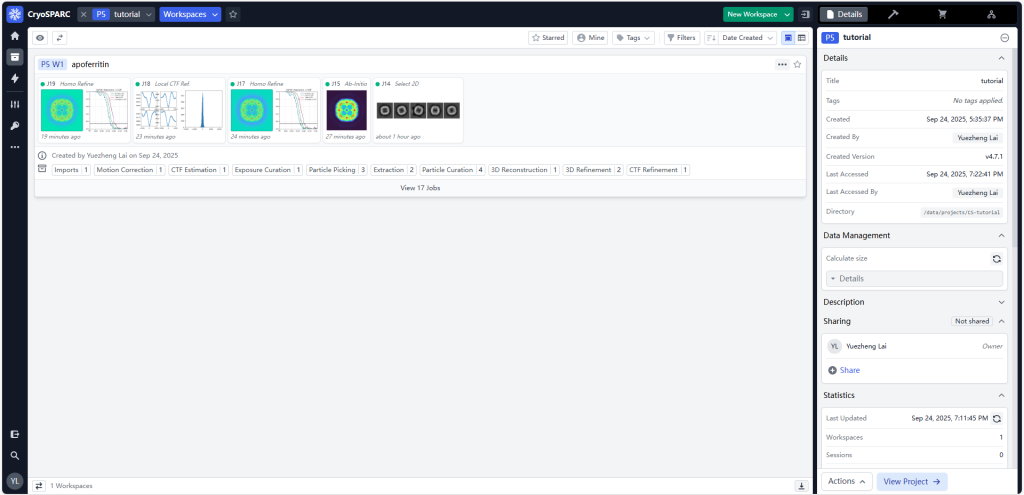
右上角Details显示Workspace或者Job的信息,锤子形状的按钮是Builder,可以新建所有类型的Job。
导入数据
冷冻电镜收集的原始图片一般是以Mutiframes Movies形式(比如tiff、eer格式)。点击Builder,在里面选择Import Movies。
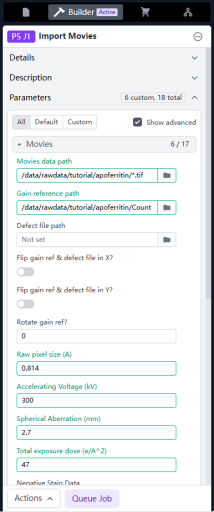
点击Movie data path右边的文件夹图标,找到自己的原始数据位置,选择其中一张照片,然后在路径里改成*.tif或者*.eer。当前目录下全部Movies都被选中了。点击Select。注意:*代表该文件夹下全部文件,为了防止一起导入当前路径下有gain或者其它文件,写*.tif保证导入的都是以tif后缀的Movies。如果文件分布在许多子文件夹内,也可以用*来代替文件夹,比如EPU软件默认格式收回来的数据,可以类似输入:
/path/to/Supervisorxxxx/Image-Disc1/*/Data/*.tif
然后,点击Gain reference path右边的文件夹图标,选中gain文件,这里是CountRef_26_000_Oct04_16.13.54.mrc。
CryoSPARC能用.gain或者.mrc后缀的gain文件。如果收集回来的gain文件是.dm4格式的,可以用eman2的e2proc2d.py脚本转化格式。
e2proc2d.py gainfile.dm4 gainfile.mrc
然后将电镜收集参数填完(这些参数在收集冷冻电镜数据的时候就要记下来)。
Rotate gain ref? 一般我们收的数据gain flip和rotate都不用管,都是正的。这套tutorial的数据需要旋转90度。输入1代表旋转一次90度。
Raw pixel size (A) 像素大小,这套是0.814,根据自己的数据更改。
Accelerating Voltage (kV) 加速电压,一般是300 kV。
Spherical Aberration (mm) 球差系数,一般生物冷冻电镜是2.7 mm,也有装了球差矫正器的是0.001 mm。
Total exposure dose (e/A^2) 总电子剂量,这套是47,根据自己的数据更改。
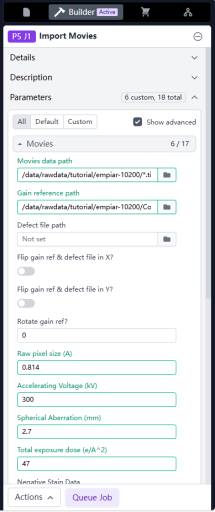
填好参数后点击点击Queue Job然后点击Queue。
如果是在别的软件或者预处理中已经做过Motion Correction的单帧mrc格式的显微照片,则使用Import Micrograph的任务而不是Import Movies,也是正常填入参数,然后跳过下一步漂移矫正,直接到CTF estimate的步骤。
漂移校正
点击Builder并选择任务Patch Motion Correction。
然后点击刚刚的Import Movies那个Job卡片的左上角,这时会显示Import Movies这个Job的详细信息,右边是MotionCorrection Job的Builder信息。
鼠标左键按住Import Movies Job右侧Output Groups中的Import Movies,此时右侧的Input框会变绿,拖住Import Movies并放入Input。
这种拖动操作在CryoSPARC中十分常用,下文不再重复。
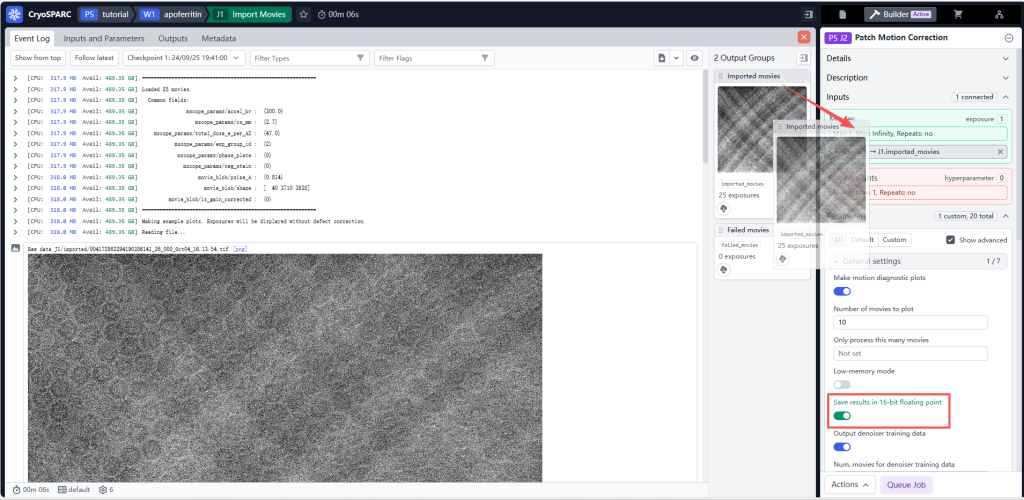
自CryoSPARC-v4.0之后新增了一个Save results in 16-bit floating point的功能,将原来的float32结果保存为float16,从而节省50%的存储空间并加快运算速度,同时对结果几乎没有影响,勾选上。
点击Queue Job然后点击Queue。
计算CTF
点击Builder并选择Patch CTF Estimation。
类似于上一步,点开Motion Correction的结果,将其OutputGroups中的Micrographs作为Input拖入Patch CTF Estimation的Inputs框内。参数默认就行。
Queue Job。
筛选照片
在数据收集过程中可能会有一些冰污染严重、像散高、位置偏移等问题的照片,可以筛选剔除,保证后续计算质量。
点击Builder选择Manually Curate Exposures。将CTF estimated的Micrographs作为Input Exposures。Paticles那一栏可以不管,它写着Min:0就是允许没有Input。参数默认。
Queue Job。
可以注意到在Builder页面中,Manually Curate Exposures右侧有一个的Interactive标签,Queue后就会显示成”waiting”的粉红色状态。这种Job就是需要交互的手动Job。点击Job左上角打开详细信息。
打开后会显示如下的交互界面。右边有三个选项Thresholds、Micrograph、Diagnostics。
Thresholds可以通过调整阈值来批量选择、丢弃照片。Micrograph会显示单张照片。一般电镜照片都是成千上万张,所以大部分时候会用Thresholds快速筛选明显有问题的照片。一般可以用CTF fit resolution和Astigmatism进行筛选。
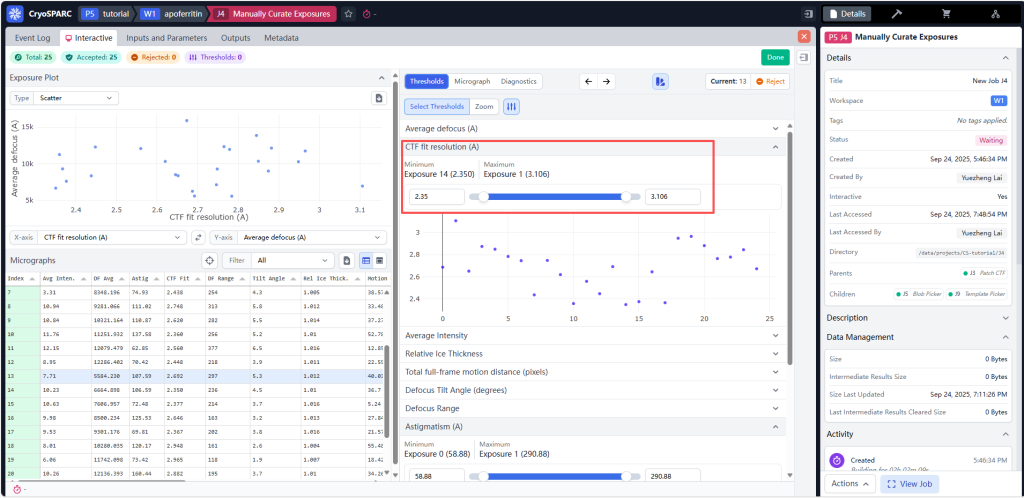
点击右上角Done完成。
颗粒挑选
颗粒的挑选以及分类往往决定了后续三维重构、分类、细化的质量和结果。挑选颗粒是选出颗粒的坐标,然后将其从一张大的照片中以一个小框扣出来。一般常用的有Manual Picker手动挑选,Blob Picker通过给定颗粒大小自动挑选,Template Picker以给定的二维模板来挑选颗粒。CryoSPARC中还有一些别的颗粒挑选的方法,比如通过深度学习训练等新的方法。
Blob Picker非常简单,直接给颗粒大小就自动挑选,它会创建一些直径的圆并作为颗粒模板。点击Builder并选择Blob Picker。将Manually Curate Exposures里accepted的Micrographs作为Input。并且在Minimum particle diameter和Maximum particle diameter参数里给出颗粒的大小范围(单位是Å)。如果你的颗粒不规则就要考虑到不同方向的投影可能大小不同。
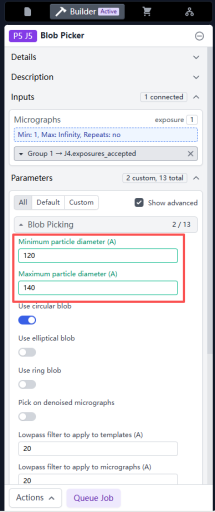
Queue Job。
Pick的结果有的时候不一定尽如人意,有大量的坐标都是选择了空的位置或者碳膜上。
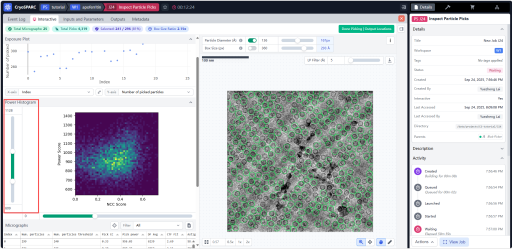
我们可以在这里用Inspect Particle Picks这个任务来调整一下。创建Job,然后将Blob Picker的结果输入进去并Queue。
可以看到在左下角有一个可以拖动的条Power Histogram,用来设置阈值。阈值的低值往上拉可以将大量空的位置筛选掉,阈值的高值往下拉可以将信号特别强的冰污染位置去掉。点击Done Picking。
颗粒提取
Pick输出的只是一个XY坐标,要后续进行计算需要颗粒的照片,需要通过这个坐标以及给定一个正方形框Box将颗粒提取成一张张小的照片。
点击Builder并选择Extract From Micrographs。
前面Inspect Particle Picks结果里有两个Output Groups:Micrographs和Particles。分别作为Extract From Micrographs的Input Micrographs和Input Particles。
然后修改Extraction box size。这个参数代表提取颗粒的box大小,至少需要比你的颗粒直径大。比如已知铁蛋白的大小是120 Å左右,pixel size是0.814 Å/pix。那么给个256 pix的box大小就足够了。注意单位,用你的pixel size来进行pix和Å之间的转化。
将Save results in 16-bit floating point勾选上。能节省一半的存储空间并提高计算速度。
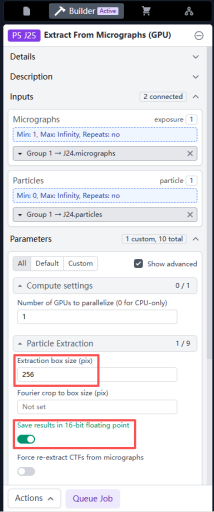
Queue Job。
二维分类
点击Builder并选择2D Classification。将Particle Extraction的Output-Particle Extract作为Input输入。参数Number of 2D classes是要分的类数目。默认是50,如果颗粒多的话可以增加分类的数目来获得更好的效果,但是也会增加计算时间。这里我们所有参数都默认。
Queue Job。
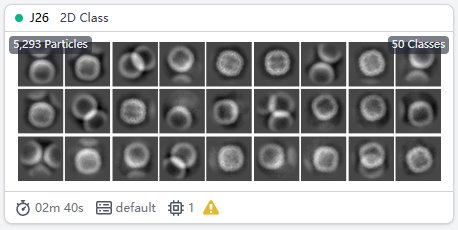
点击Builder并选择Select 2D Classes。将刚刚的二维分类2D Classifications的两个Output(All particles和2D class averages)作为Input输入。
Queue Job。这也是个Interactive Job。
选择自己想要的二维平均,点击Done。
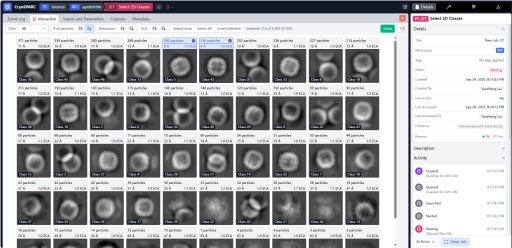
在实际的数据处理过程中,往往一轮二维分类无法达到好的效果。挑选好的二维类,然后用Selected Particles作为输入再进行二维分类。循环直到达到满意的效果。
用模板挑选颗粒
理论上有了Particle stack,就可以进行三维重构了。这里再展示另一种挑选颗粒坐标的方法。
刚刚挑选了比较好的二维分类结果的模板,就可以进行Template Picker了。点击Builder并选择Template Picker。
将刚刚Select 2D Classes里Output-Templated Selected作为Template Input。
将Curate Exposure-accepted作为Micrograph Input。
然后输入颗粒大小Particle Diameter(铁蛋白120Å)。
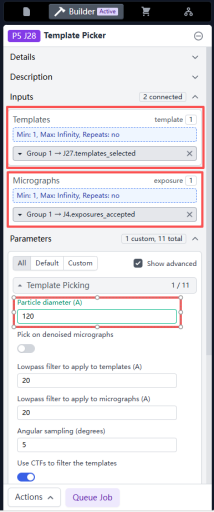
Queue Job。
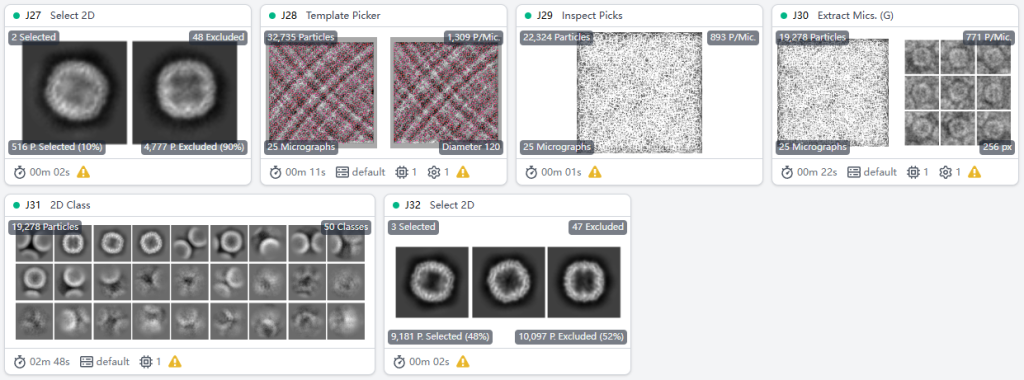
然后和之前步骤类似,用Inspect particle剔除假阳性颗粒,提取颗粒Extract Particles,二维分类2D Classification,选择分类Select 2D Classes。
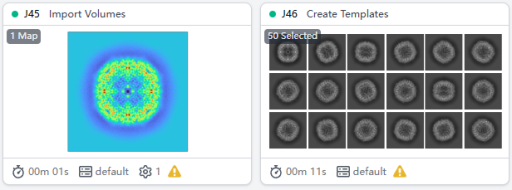
除了这种挑选颗粒生成2D template,还有另一种方法生成template。如果你之前已经有了这个蛋白类似的3D map。点击Builder并选择Import Volume。然后用Create Template任务可以将3D Volume进行2D投影,从而生成2D Template。这样就可以跳过耗时的通过初始二维分类直接获得template。
三维重构
Ab-initio可以从Particle stack从头开始生成初始三维模型,后续再用这个模型可以用来三维分类或者细化。
点击Builder并选择Ab Initio Reconstruction。将最后一轮Select 2D Classes作为Particle stacks输入。
Number of Ab-Initio classes可以填入想要生成几类不同的map,Ab-Initio随后Heterogeneous refinement本身也是三维分类的方法之一,可以分出构象差别较大的类,剔除二维分类中没有用的杂颗粒。
由于这套数据是模板,颗粒均一性很好,所以不需要三维分类也能获得好的结果,因此Number of Ab-Initio classes默认1就行。
铁蛋白是高度对称的球体,我们在Symmetry给个对称性O。
Queue Job。
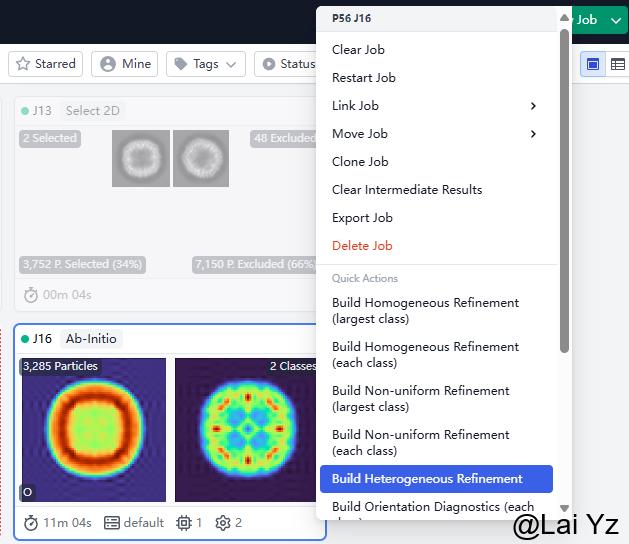
如果Number of Ab-Initio classes不止一类,点击Builder并选择Heterogeneous Refinement。将Ab-intio结果中每个class的Particle和Volume都拖进Input中,至少需要两个Volume来进行分类。现在CryoSPARC中有Quick Action的功能,鼠标右键点击Ab-intio的结果,会有一个Build Heterogeneous Refinement的选项,一步到位。其他的任务右键点击也有自己的对应的快捷创建任务,可以都去试试。
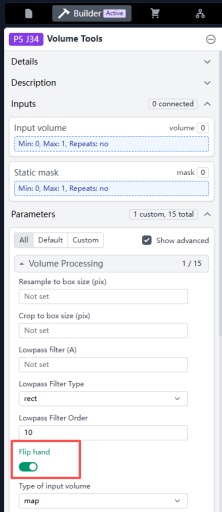
注意!在Ab-intio从头生成3D map过程中,可能会出现生成的map手性错误的现象,需要手动检查,可以用Volume Tools选Flip hands可以将Volume的手性翻转。然后用正确手性的map替代掉那个错误的map来进行后续步骤。
Refinement
常用的有两个Job,Homogeneous Refinement和Non-uniform Refinement。Non-uniform Refinement是对小蛋白和膜蛋白是可以有更好的优化效果。
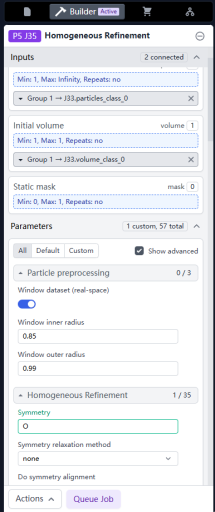
这里选择Homogeneous Refinement,一般速度会比Non-uniform refinement快。
Input需要一个particle stack和一个volume,这里将Ab-initio的颗粒和volume作为Input。
如果前面提到的Ab-initio不止一类并进行了Heterogeneous Refinement,选择Heterogeneous Refinement结果中较好的class,Particle stack颗粒集和对应的Volume map作为Input。
同样Symmetry给个O对称性。
由于铁蛋白本身是个标准蛋白,又给了高对称性,所以很容易就到了2.5A的高分辨率。
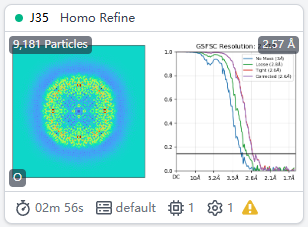
点击Output里一个云和一个箭头的图标可以下载结果。
map就是获得的密度图。map_sharp是sharpen后的map,在job的详细信息Guinier Plot里可以看到map sharpen B Factor。map_half A/B是half map。现在上传pdb数据库已经必需上传half map了。
局部CTF优化
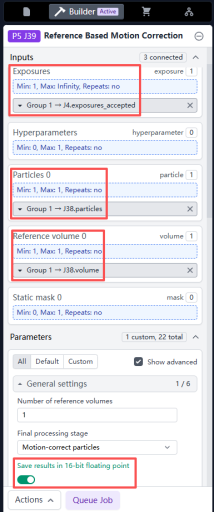
Local CTF Refinement会对每一个颗粒的局部CTF做一个校正,有可能进一步提高分辨率。将Refinement的Particle和Volume输入Local CTF Refinement。然后将Local CTF Refinement输出的校正过的Particle stack和前面Homogeneous Refinement的Volume作为输入重新做一次Homogeneous Refinement。在实际运算过程中这一步往往都能提高不少分辨率。
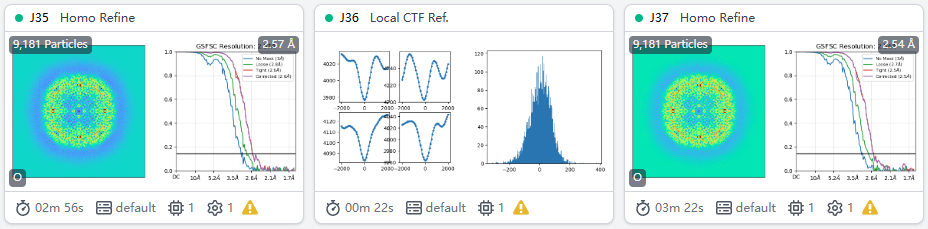
Reference based motion correction
在CryoSPARC v4.4之后,CryoSPARC推出了一个新的Job叫Reference Based Motion Correction,它也是一种Motion correction的方法,但是是根据最后Refine完的颗粒的先验姿态信息,来更精确地对每一个颗粒进行Motion correction。这个方法类似于RELION中的Bayesian Polishing,加上CryoSPARC自己的超参数优化及GPU加速等方法。
重新做一次Refine,并勾选上Minimize over per-particle scale。

选择Reference Based Motion Correction,将Refine的Particle和Volume作为输入,将前面做的Curate Exposures(如果没有做筛选,就是Patch CTF estimation)的Micrograhs作为Exposures那栏的输入。这三个输入是必须的。照常勾上Save result in 16-bits floating point,然后Queue。
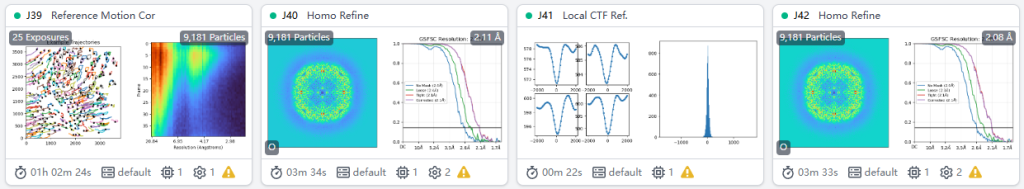
将这一步的输出的颗粒和之前Refine的Volume再重新做Refine > Local CTF refine > Refine。我个人尝试下来对于3 Å左右的实际数据大多能提高0.1-0.2 Å左右。在这套铁蛋白数据中分辨率最终达到了2.08Å。
评估结果
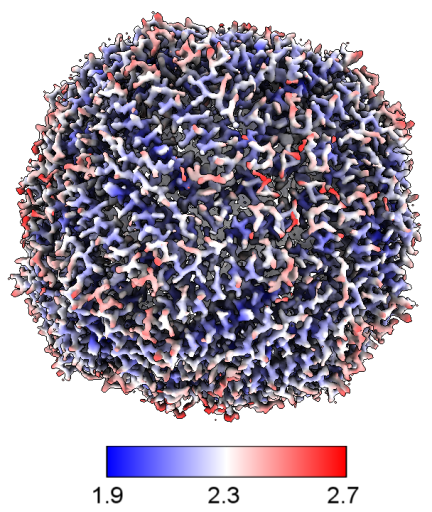
发表文章中可能需要一些数据和图片来对结果进行评估。比如Local resolution,将最终Refinement的结果输入。在结果中选择那个叫做map_locres的下载,在ChimeraX等软件中可以展示。
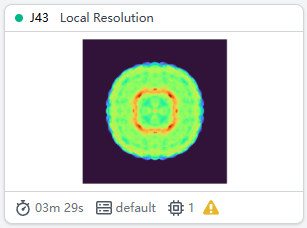
下载后同时打开map_sharp和map_locres。上方工具栏选择Tools > Volume Data > Surface Color。弹出工具栏后Color surface选择map,选择by volume data value,using map选择locres。颜色输入对应分辨率。点击color。
CryoSPARC v4.4以后的新版的CryoSPARC将ThreeDFSC这个Job删除了,但是新推出了Orientation Diagnostics。将最终Refinement的Volume作为输入就可以。CryoSPARC v4.5版本之后的样子:
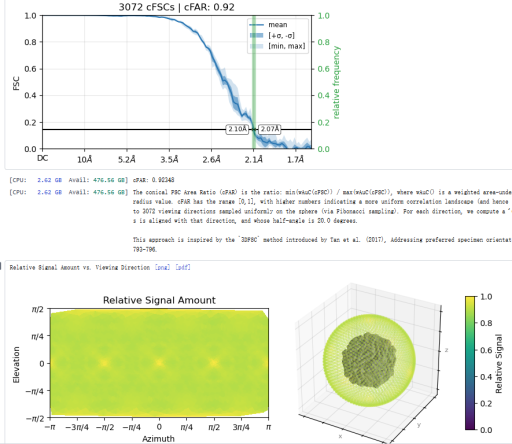
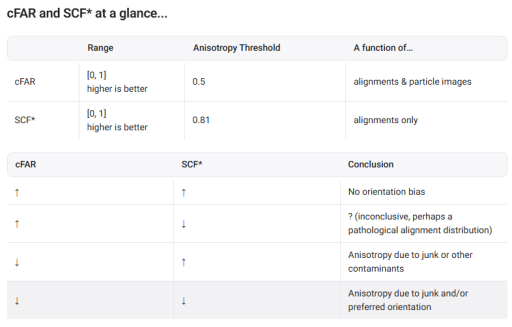
根据官网给出的建议,综合考虑cFAR和SCF来判断数据质量,cFAR(the conical FSC area ratio)数值小于0.5以及SCF小于0.81应该要考虑取向优势的问题了。
lai Yz您好,我们这边学习结构计算时,在exrtaction的Extraction box size (pix)设置后,还会设置Fourier-crop to box size (pix)为4的倍数,如64,但远小于Extraction box size (pix),这样可以大幅度减少内存和提高计算速度。等Ab分出比较好的类后再提高Fourier-crop to box size (pix)或者不设置,使用bin1来进一步提高分辨率。
可以的,实际数据的计算中基本上前期都会bin2或者bin4能大大加快运算速度。
作者您好,我是计算机专业做交叉研究的同学,想请教您一个概念问题,bin4是否就是Output F-crop factor设置为1/4,bin2是否就是设置为1/2
是的
您好,我想把两次300kV收集的数据合并处理,是在两台电镜收集的相同的样品,请问可以合并吗?有没有简单的操作流程?参数设置需要注意啥?是要两套数据分别提取颗粒后在合并吗?
两个都重新提取颗粒,其中一组提取的时候crop一下,使得最后的pixel size相同,cryosparc应该对pix的要求是差距在0.0001Å。找一个合适的box和crop box。
作者您好,我使用Reference Based Motion Correction后发现particles的box size由原本的240变成了480,请问您有遇到这个情况吗?我只能重新extract particles设置box size为240
是不是super-resolution模式收集的,你在做motion correction的时候给bin2了?试试fourier crop to box size这个参数给240。
是的,感谢解答,现在box size是正确的。
我发现虽然box size和pixel size和使用reference based motion correction前一样,但是两个map比较时无法调到level值一样去比较,有点困惑。fourier crop to box size这个参数给240比不给然后重新extract最后得到的map质量要好一些。
cryoSPARC中 使用mask在local refine中 怎么样才能分出mask部分更多的状态呢
可以用3D classification,给一个local focus mask
您好,我想导出workflow,但根据官方教程选择要导出的job后,没有“create workflow”选项,该怎么办呢
不清楚,可能是版本问题,我这都是有的