下面列了几种在搭建结构后进行评估时需要着重关注的几个点。
Peptide Bond Validation
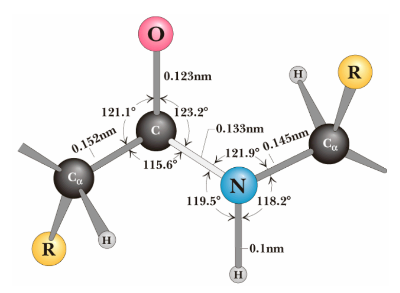
很好理解,键长键角的参数。在蛋白质中,C-C键的键长约1.52Å左右,肽键的键长约1.33Å,C=O键的键长约1.23Å。
Ramachandran Plot
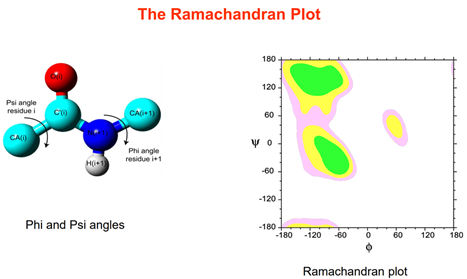
拉式图(Ramachandran Plot)是经典的评估蛋白质主链构象是否合理的方法。
众所周知,肽平面是由构成肽键的四个原子(C、H、O、N)以及相邻两个Cα这六个原子组成的。由于肽键有一定双键性质,因此肽平面是刚性的,它们一定在一个平面上。
围绕Cα和N键的旋转角叫做φ(phi),围绕Cα和C键的旋转角叫做ψ(psi)。
虽然理论上的C-C单键可以任意旋转,但是由于空间位阻,ψ和φ并不是可以取任意值的。
生物物理学家Ramachandran等人构建了模型,计算得到了ψ对φ立体上允许的值,就是拉式图。
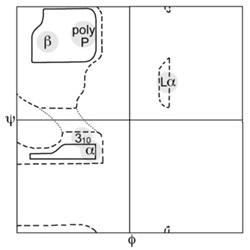
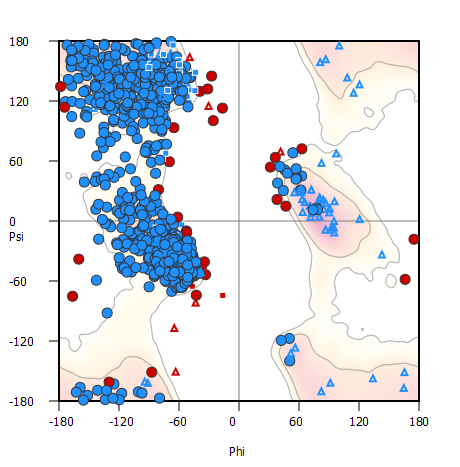
拉式图的不同区域表示了不同的二级结构。β是β折叠,α是α螺旋,310是310螺旋,Lα是左手螺旋。
随着不断有高分辨的蛋白质结构被解析,更加精确的拉式图也不断更新。
Ramachandran plot outlier一般表示,以高质量蛋白质结构为参考,构象存在可能性<0.5%的主链构象。在拉式图中展现出来就是在封闭轮廓外面。
一般来说搭建结构的时候,Favored的氨基酸至少要占蛋白质90%以上,Outlier越低越好。
多数氨基酸的拉式图是类似的,有一些氨基酸是特殊的。比如Glycine甘氨酸,它的侧链只有一个H原子,范德华半径更小,因此限制较小,在拉式图中Allowed的范围就更大。而Proline脯氨酸Cα和N成环,限制较大,在拉式图中Allowed范围就小。
下面就是一个真实的结构用Coot计算出的拉式图,三角形代表了Gly,正方形代表了Pro。
Rotamer
氨基酸侧链可以有多种不同的构象,但是每一类型的氨基酸都有几种比较稳定的构象。
Rotamer outlier一般表示,以高质量蛋白质结构为参考,可能性<0.5%的侧链构象。
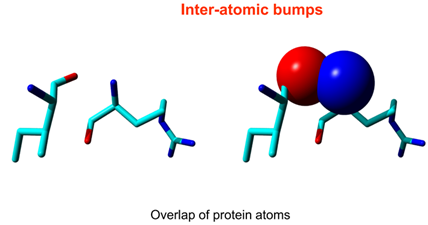
Clashscore
Clashscore是描述原子间冲突的参数。一般而言,包含H原子的情况下,原子原子间overlap大于0.4A的被认为存在较严重的clash。Clashscore表示为每1000个原子中存在clash的原子数量。
搭建电镜结构最好把clashscore降低到10以下。
一般有严重clash的地方往往结构上也存在其它问题,最好确认问题出在哪里。
如果确认不是结构搭建的有问题,clashscore太高的话可能要考虑下pixel size是否有问题,用Calibrated pixel size重新计算map或者做个post processing。
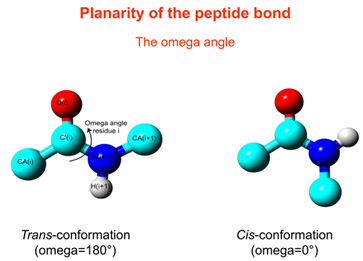
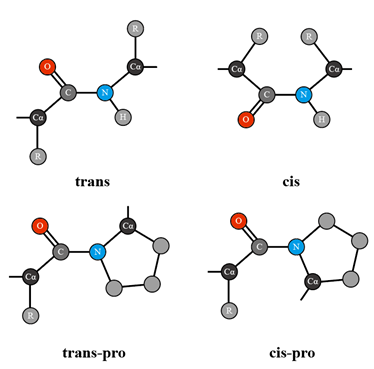
trans/cis peptide
C=C双键存在顺式(trans)和反式(cis),肽键有部分双键的性质,也类似有顺反式构象。顺式肽平面中两个Cα总是在两侧的,蛋白中的氨基酸大多数是trans构象,也就是omega角度为180°。但是也有少数cis构象的氨基酸,omega为0°。
尤其是有Proline的位置,cis-pro出现的概率相对别的氨基酸高一点。
至于Cis-nonPro,出现的概率大约1/3000,在分辨率足够好(<2.5Å)或者有同源结构参考的时候可以谨慎搭建。
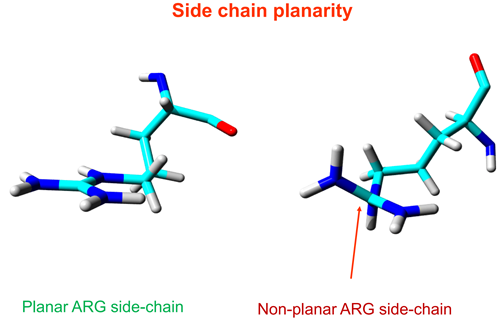
Side chain planarity
同样,由于空间位阻和立体化学,一些大侧链本身内部也是存在一些限制的,比如Arginine的头部的胍基。
Reference:
Luigi Vitagliano. Protein structure validation and quality assessment (2019).