在处理电镜数据的过程中,由于蛋白质往往存在柔性和刚性的区域,会遇到某个关键的区域分辨率不佳的情况,这可能是由于该区域柔性相对较大导致的。
这时候通过局部细化Local Refinement在许多情况下能很好的解决问题。
Local Refinement需要创建一个Mask包裹想要细化的区域,以及另一个Mask包裹需要减掉的区域。在目标区域内进行Refine获得更高局部分辨率。
这里参考CryoSPARC官网的一套Tutorial,用的是EMPIAR-10073的一套酵母U4/U6.U5 tri-snRNP的冷冻电镜数据,是已经经过初步的分类将坏颗粒剔除掉了的Particles数据集,可以直接在EMDB数据库下载,约70G左右。
附上官网教程:
下载并导入数据
用wget命令从EMDB数据库下载数据。
wget ftp://ftp.ebi.ac.uk/empiar/world_availability/10073/data/*.mrcs
以及对应的STAR文件。
下载后会有后缀为mrcs的particle images和后缀为star的文件,在CryoSPARC中Build>Import>Import Particle Starck。
在Particle meta path参数中选择下载的shiny_correctpaths_cleanedcorruptstacks.star(官方Tutotial里给的star文件有几个对应的images缺失了,如果报错,用文本编辑器打开star文件删除对应的条目即可)。
在Particle data path参数中选择存放数据的目录。
注意在star文件中写的索引路径是Particle/*.mrcs。因此需要在工作目录(我这里是/data/testdata/10073)下建立一个名为Particle的目录并把数据放进去,这样根据star文件索引的时候才能搜索到数据。
其余参数默认。
Refine和Local Resolution
这套数据由于已经经过了初步分类,可以在一切默认参数下直接运行Ab-initio加上Homogeneous Refinement就可以达到3.6Å左右的分辨率。柔性较高的结构用NU-refinement会好一些,能到3.55Å左右。
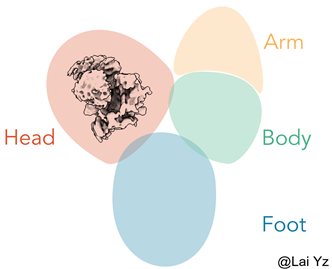
我们会发现这个结构中心区域局部分辨率明显较高,而头部和尾部分辨率基本大于6Å。
创建mask
接下来需要在ChimeraX里创建一个包裹想要进一步细化区域的Mask和一个包裹其它需要减去区域的Mask。
打开NU-Refinement生成的Map。先用高斯滤波将Map平滑。平滑的mask很重要,没有低通滤波的mask包含高分辨率信息,后续的refinement可能会仅根据mask而不是volume本身在half map之间产生虚假的相关性,这会导致虚高的GSFSC,所谓的overfitting。
volume gaussian #1 sDev 2
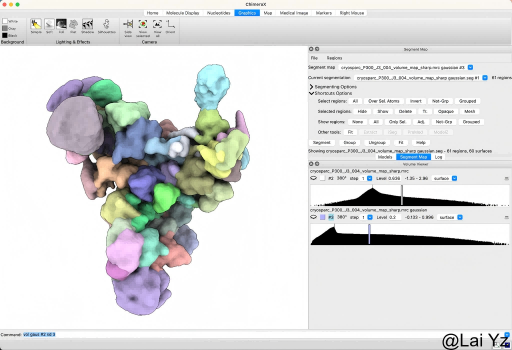
然后使用Segger工具将volume分区。在ChimeraX中点击Tools - Volume Data - Segment Map。在弹出的框中选择刚刚生成的高斯滤波的volume,然后点击Segment。
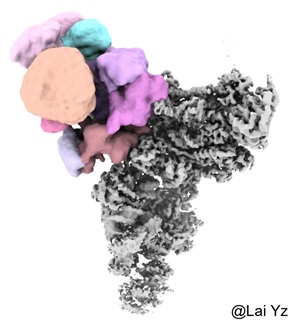
map已经被分成了多个区域,现在需要做的是选中头部区域的块,操作详见ChimeraX使用。
Ctrl+Shift+鼠标左键可以连续选中区域,然后点击Group可以把多个区域合并成一个区域。点击Hide/Show/Delete就是对应的隐藏/显示/删除。差不多达到这个效果:
这样生成的volume的box size是不对的,我们需要把它调成和我们原始map相同。这里#4是要保存的mask,#1是最开始的map。
volume resample #4 ongrid #1
这样就会生成一个新的#5的map,保存它,然后上传到服务器上。
扩张和填充
回到CryoSPARC,选择Import 3D Volume。参数默认,把上传的map给import进去。
然后再创建Volume Tools,将刚刚import的map输入进去,参数上Type of output volume选择mask,Threshold选择一个在ChimeraX中打开看着能覆盖区域的Contour level,这里给0.05,Dilation radius和Soft padding width就是扩张和填充,分别给5和17。这个job将会将map二值化为mask,同时向外扩张5个pixel,同时用17个pixel的软边缘填充。填充半径大一些会保证mask的质量,但是太大会导致对感兴趣的区域的对齐效果下降,可以从大了往下试。默认值是13。
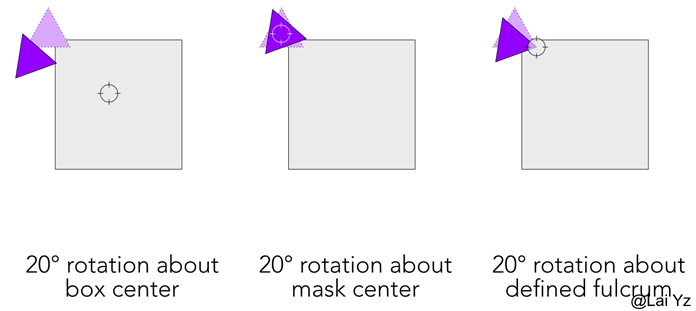
寻找支点(可选)
CryoSPARC的Local Refinement允许指定感兴趣的Volume可以围绕其旋转的”支点”。使用支点的位置坐标,CryoSPARC将重新计算局部角度搜索网格,使其更加准确。这相当于在每个粒子图像中将粒子重新定心到支点的位置。一般情况下这个差别不会很大,平时可以先直接省略支点,默认效果很差再考虑这个。
默认情况下是围绕mask的中心旋转。当mask覆盖总volume的很大一部分时,这样效果会比较好。这个教程里头部区域没有覆盖volume的很大一部分。在这种情况下,最好围绕box的中心旋转粒子,因为这更接近真正铰链运动。当mask覆盖中等大小volume时,围绕box中心的旋转往往效果更好。没有一个最佳答案。直觉告诉我们,头部应该围绕头部和身体界面上的某个点旋转。
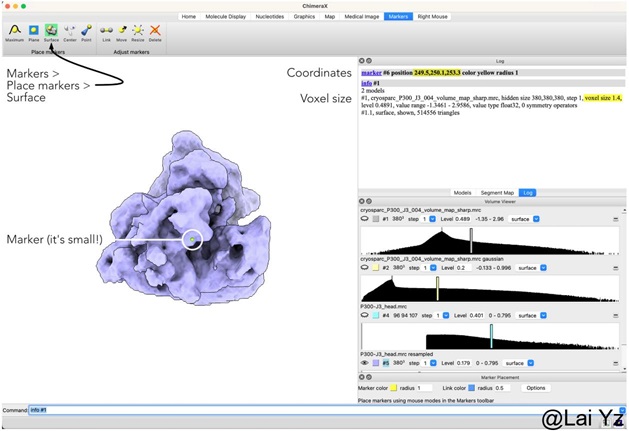
我们可以用ChimeraX找到这个点。打开ChimeraX,下载这个生成的mask。在上方工具栏找到点击Markers - Surface,然后在差不多的旋转点上右键点击一下。在右侧日志栏就会显示这个点的坐标。注意单位是Å,要换算成pixel,除以pixel size就行。输入info #1可以显示。
在后续的Local refinement任务里把这个坐标输入到fulcrum就行。
Particle Subtraction(可选)
Particle Subtraction从NU-Refine或Homogenous Refine中获得的整体结构,并从原始Particle images中减去其Mask区域的投影,创建一个Subtract后的Particle stack。这些Particles可以用于Local Refinement,现在减去了不需要的信号,对齐将仅基于Focused区域,获得更好的对齐。
在ChimeraX中类似于上面创建头部mask的过程,这次创建不想要区域的mask,后处理的步骤与前面一致,但是在扩张和填充时,我们不希望把周围的信号都减掉,只希望减掉主体蛋白信号,因此扩展参数dilate radius可以不要,但是padding是必须的,给到推荐最小值12(5*resolution/apix)。
导入并处理好mask后,选择Builder - Local Refinement - Particle Subtraction。将前面做的NU-Refinement的Particles和Volume作为Input,把处理过的包括除了头部Mask之外所有区域的Mask作为Input。这样就可以生成一个新的Particle stack数据集,只留下了头部的信号。
这时候如果做一个Homogeneous Reconstruction Only的任务,我们很快就能看到其余部分被减掉了。
Local refinement
然后选择Local Refinement。用刚刚Subtract的Particle Stack作为Particles Input,NU-Refinement的Volume作为Volume Input,处理过的头部Mask作为Static Mask Input。如果没有做Substract,直接用NU-Refinement的particle就行。
最终能获得一个3.8Å左右的head局部分辨率。
合并
可以用ChimeraX的vop maximum命令或者Phenix的Combine map把局部map和整体合并。