总结几个做mask的方法。
这里用一个ATPase作为例子,它分为可溶区头部和一个跨膜区,这里打算做一个跨膜区的mask做local refinement。
橡皮擦
这是最简单直接的方法,用Chimera或者ChimeraX打开map,然后用Tools > Eraser直接一点点擦就好了。
用PDB结构生成
在ChimeraX打开结构,#1是map,#2是model。
先将pdb模型fit到对应位置,用molmap命令生成map。
molmap #1 20 onGrid #2
#1是pdb,#2是map,20是分辨率可以酌情调整。但是不能太小,最好大于10 Å。
然后保存新生成的#3 map,就可以在cryosparc里import volume了,记得type选择是mask,做一下柔化啥的,和之前的Local refinement教程里一样。
Map segment
这是贴在里的方法,也统一放在这页里,来自CryoSPARC Guide,使用Segment方法分区,然后选区。
打开的Map。先用高斯滤波将Map平滑。平滑的mask很重要,没有低通滤波的mask包含高分辨率信息,后续的refinement可能会仅根据mask而不是volume本身在half map之间产生虚假的相关性,这会导致虚高的GSFSC,所谓的overfitting。
volume gaussian #1 sDev 2
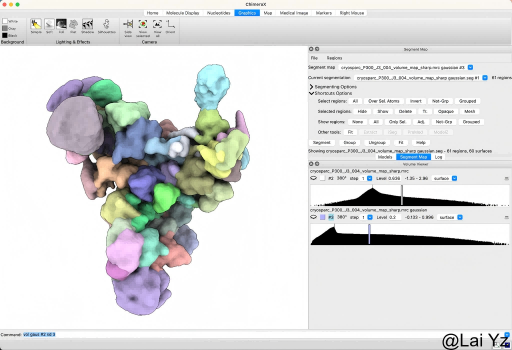
然后使用Segger工具将volume分区。在ChimeraX中点击Tools - Volume Data - Segment Map。在弹出的框中选择刚刚生成的高斯滤波的volume,然后点击Segment。
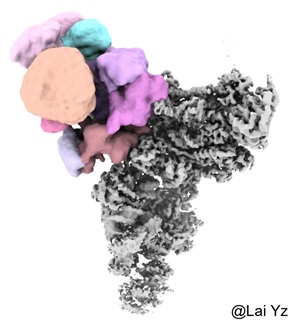
map已经被分成了多个区域,现在需要做的是选中头部区域的块。
Ctrl+Shift+鼠标左键可以连续选中区域,然后点击Group可以把多个区域合并成一个区域。点击Hide/Show/Delete就是对应的隐藏/显示/删除。差不多达到这个效果:
这样生成的volume的box size是不对的,我们需要把它调成和我们原始map相同。这里#4是要保存的mask,#1是最开始的map。
volume resample #4 ongrid #1
这样就会生成一个新的#5的map,保存它。