RELION(for REgularised LIkelihood OptimisatioN, pronounce rely-on)是经典的冷冻电镜数据处理软件包,目前已经出到5.0版本。相较于简单直观的cryoSPARC,RELION的UI更朴素一些,稍微复杂一些。本文用RELION4.0处理一套apoferritin的数据。同一套数据用cryoSPARC处理参见之前的教程。
如果完全不懂linux系统和命令行操作,可以先看一下Linux基础教程,这里会涉及到命令行的操作。
新建
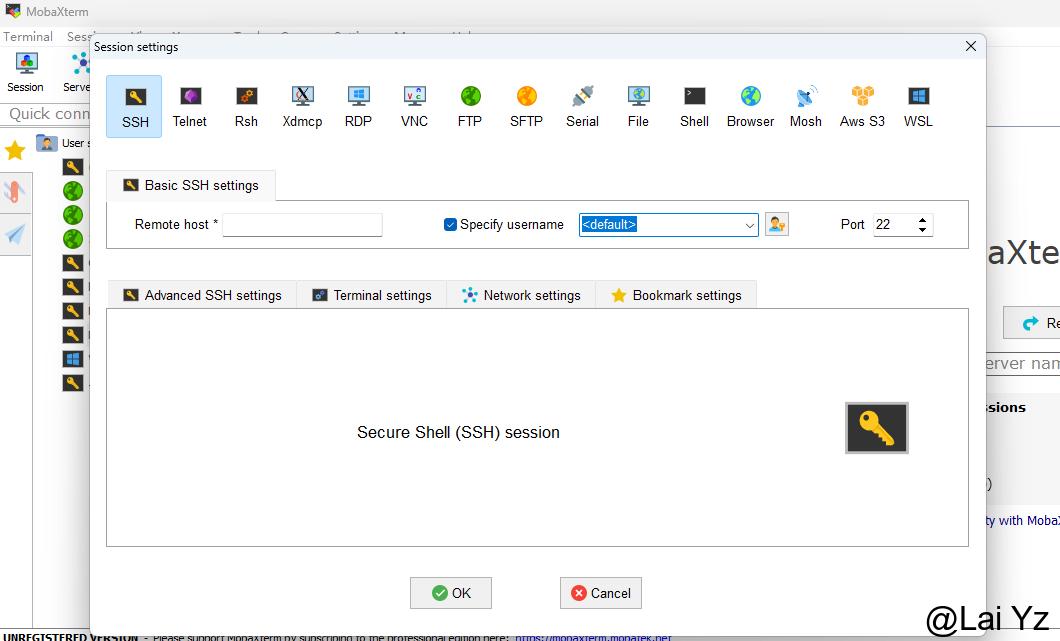
RELION需要从shell打开。我推荐下载工具,界面比较美观。点击左上角的Session然后选择SSH,Remote host输入IP地址,username输入用户名,一般端口号22是给ssh的不用改。
首先进入自己的目录下,创建一个的文件夹。输入RELION回车。如果第一次打开RELION,会提示是否新建project。点击yes。(RELION-3是命令行弹出的,输入y,回车)。
mkdir -p /data/lyz/relion_tutorial
cd /data/lyz/RELION_tutorial
relion
mkdir -p是逐级创建目录,把后面的路径换成你自己的。注意!RELION会在当前路径下新建default_pipeline.star文件和.gui_projectdir隐藏文件,下一次再在这个目录下再打开RELION识别到这些文件就不需要确认新建了。一定要在自己的文件夹下打开RELION,不要打开命令行界面就直接输入RELION。
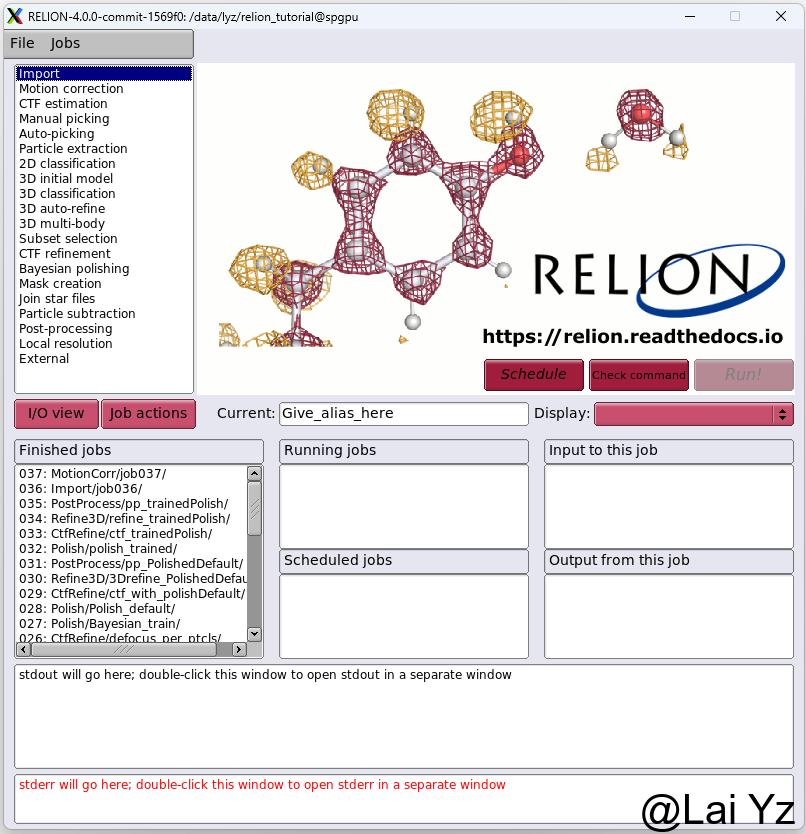
这就是RELION4.0的主界面,左侧是Job类型。
导入数据
RELION只能识别RELION目录内的文件(default_pipeline.star和.gui_projectdir所在这个文件夹下)。我们可以在RELION文件夹内创建一个rawdata文件夹,并将原始数据软链接过来。你可以理解为Windows下的创建快捷方式。这样就可以不用挪动原始数据,保证这个目录下都是计算数据。
另外开启一个shell窗口。
cd /data/lyz/RELION_tutorial
mkdir rawdata
cd rawdata
ln -s /data/testdata/apoferritin/Movies/*.tiff .
ln -s /data/testdata/apoferritin/Movies/gain.mrc .
回到RELION的界面上来,左侧选择Import。
Import raw movies/micrographs?默认yes。如果要import别的就选择no,然后选择上面的Others去导入别的文件。Raw input files输入rawdata/*.tiff
代表选中所有tiff文件。然后把Pixel size Voltage Spherical aberration等电镜参数填好。下方Current可以给Job重命名方便自己辨认,默认就是数字编号。
点击Run!
下面log框里会显示进度,如果有error或者warning会显示在再下面的log框里。
漂移矫正
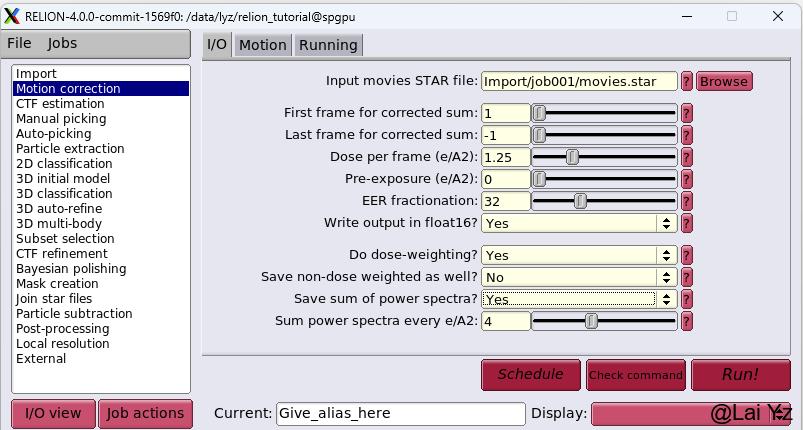
选择Motion correction,点击Input movies STAR file右侧Browse,RELION软件生成且识别.star后缀的文件,选择Import/job001/movies.star文件。
Dose per frame输入1.25。用总电子剂量totoal exposure dose 50除以照片帧数frames 40。
Write output in float16?选择yes。这是RELION-40增加的功能,相较于float32它可以节省一半的空间并增加运行速度。但是还和别的软件不太兼容比如Gctf以及MotionCor2。
Do dose-weighting默认选择yes。因为存在辐照损伤,需要对照片进行剂量加权校正。
Save sum of power spectra?选择yes。这个同样不兼容MotionCor2。
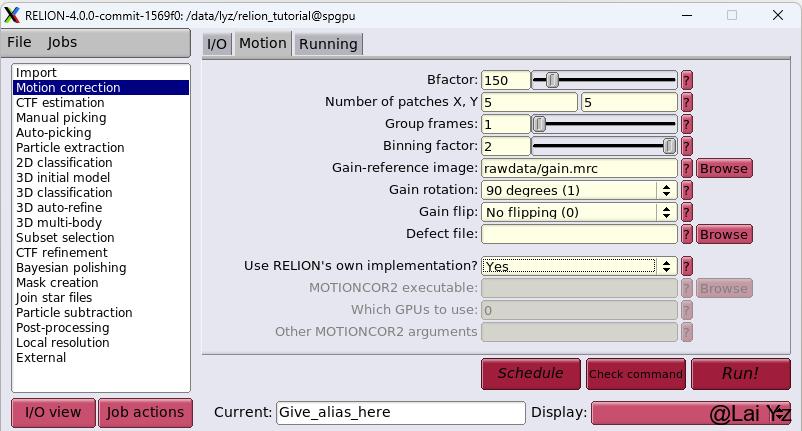
然后上面选择第二个Motion选项。
Number of patches X, Y选择5,5。这个照片是正方形
Binning factor选择2,这个照片是Super-resolutino模式收集的。
Gain-reference image选择我们rawdata文件夹下的gain.mrc。Gain rotation并且这张gain需要旋转90度。
Use RELION's own implementation?默认选择用RELION自己的算法,用的是CPU计算。也可以选择No然后选择别的软件比如MotionCor2。
Running选项卡里可以不用改,我们的示例数据很小,一下就算完了。数据多了多核(MPI)并行运算。
像Motion correction、CTF estimation、Auto-picking、Particle extraction这类Jobs,需要MPI和GPU数量一致。而2D classification、3D classification、3D initial model、3D auto-refine等Jobs,需要MPI比GPU多一个用于统筹。自己注意合理分配运算资源。
点击Run!
计算CTF及筛选照片
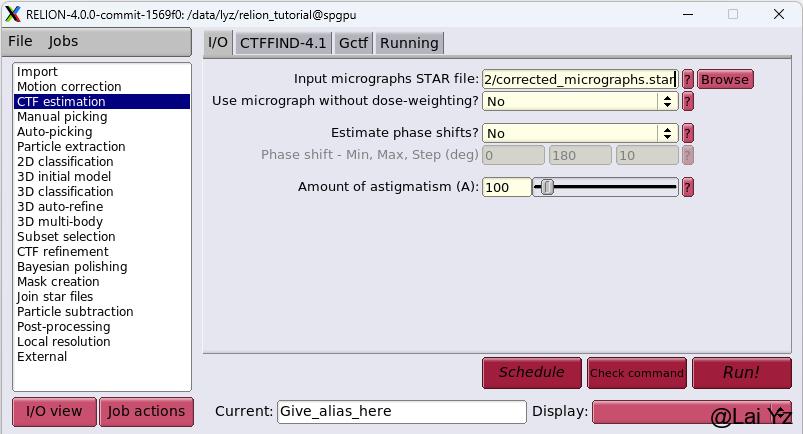
选择CTF estimation,Input选择上一步做完的Motion corrected的.star文件。
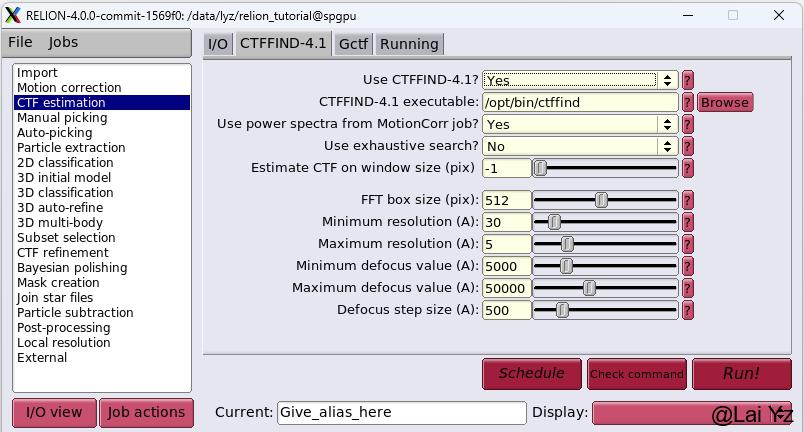
有两种CTF算法可以选择:CTFFIND-4.1和Gctf。CTFFIND使用CPU计算,Gctf使用GPU计算。这里使用CTFFIND-4。CTFFIND-4.1 executable选择CTFFIND-4.1的可执行文件,在我们服务器上这里默认的就是对的。其他参数默认。
点击Run!
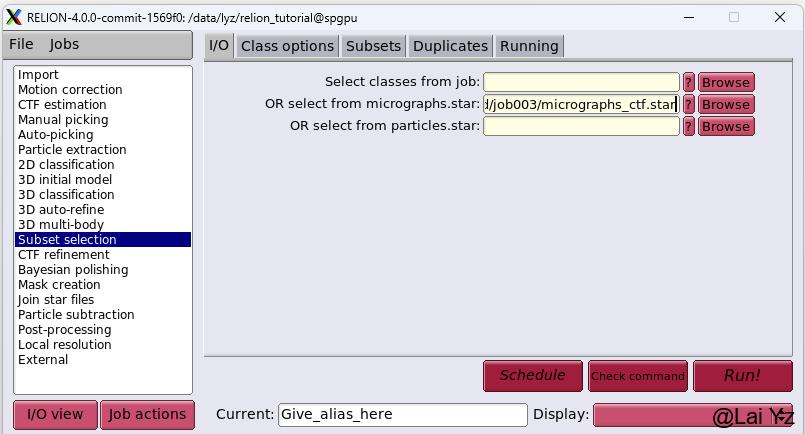
根据照片的CTF fit resolution、Astigmatism等参数可以剔除一部分不好的照片。选择Subset selection。RELION中照片、颗粒、2D、3D等的挑选都是用这个Job。在I/O选项卡中OR select from micrographs.star选择上一步的结果的micrographs_ctf.star。
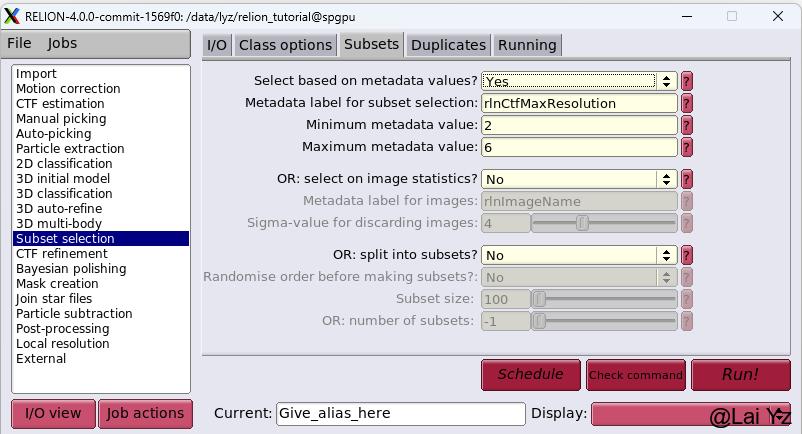
在Subsets选项卡中第一项Selected based on metadata values选择yes。将rlnCtfMaxResolution的最小最大值范围调节到2~6。
点击Run!同样的方法可以针对Astigmatism、Defocus range等筛选图片。
点击Run!。同样的方法可以针对Astigmatism、Defocus range等筛选图片。
颗粒挑选和提取
有Manual Picking和Auto Picking两种方法。先说Manual Picking。
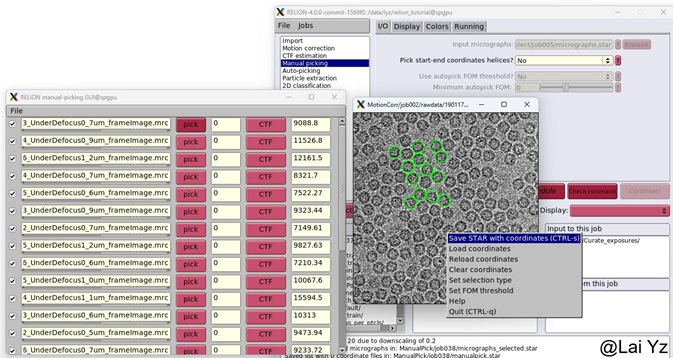
Input选择上一步Select的micrographs文件。Display - Particle Diameter可以选120Å对应apoferritin的大小,不影响结果。
点击Run! 弹出GUI。选中其中一张图点击pick。鼠标左键选中 ,鼠标中间取消,鼠标右键可以弹出保存、清除等等操作。选完了记得Save STAR file保存一下。然后关闭pick窗口。点击File-Save selection保存。关闭窗口。
然后是Auto Picking,有几种自动的方法。
Input还是选择上一步Subset select的Micrographs文件。
第一种方法是Use reference-based template-matching。需要给一个模板。
第二种方法是use Laplacian-of-Gaussian。类似Blob Picker只需要给颗粒直径范围。
第三种方法是use Topaz。用独立的Topaz软件。
只能选一种Yes,然后到对应的参数列表里改参数。Laplacian需要给颗粒大小范围。如果是Reference-based则要给2D average或者3D Volume模板。Topaz是独立软件另说。autopicking里是通用参数。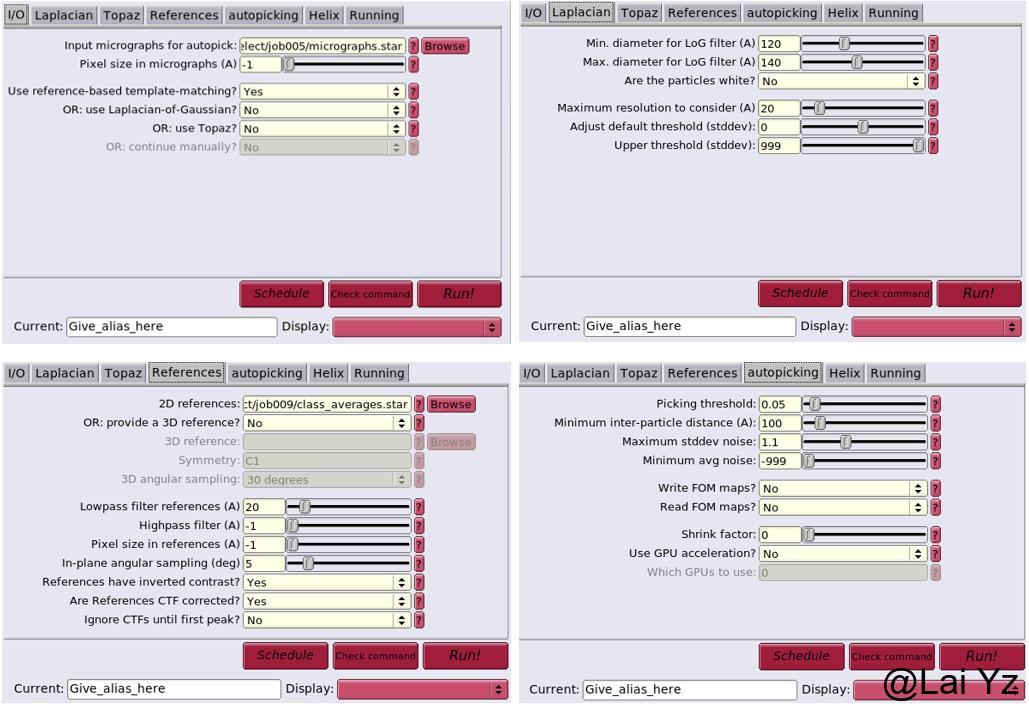
然后点击Run!
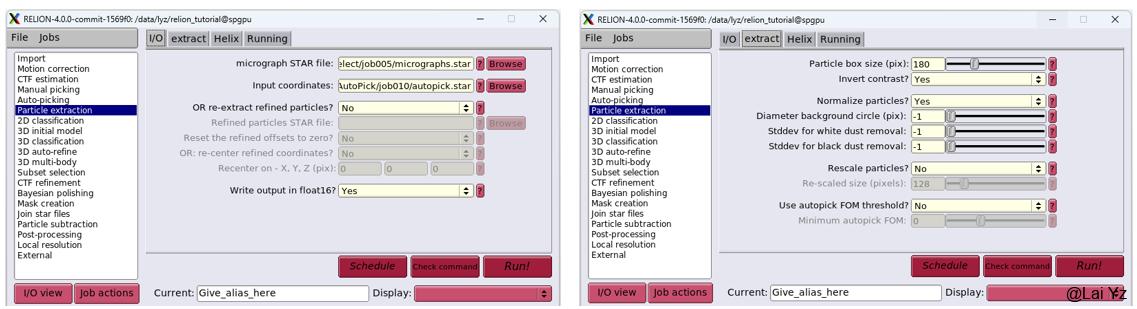
无论是怎么挑选颗粒,结束后需要提取颗粒。选择Particle extraction,Input选择Subset selection的Micrograph Subset和Manual/Auto picking的particles.star文件。Box size选择至少比颗粒大20%以上的大小。
然后点击Run!
二维分类
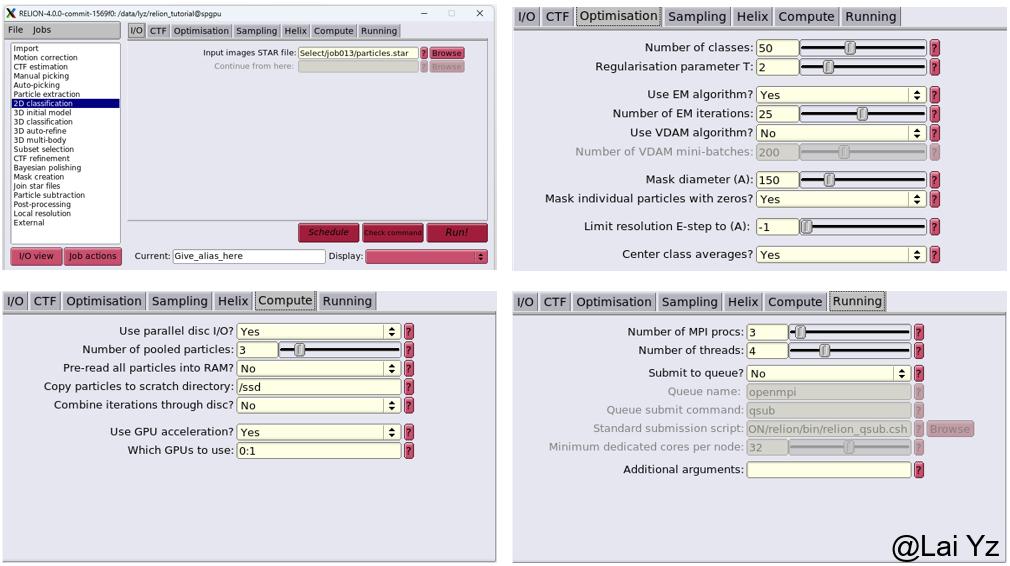
左侧选择2D classification二维分类。
Input images STAR file选择上一步Particle extration的particles.star。
Number of classes选择50类。
Regularisation parameter T这个T值代表了实验数据与先验数据之间的相对权重,也就是颗粒平均的权重更大还是实际颗粒的权重更大,大于1说明实验数据权重更大。这个值可以调整,普通情况下对于2D Classification来说1到2,对于3D Classification来说2到4,是比较合适的。太小的T值会降低分辨率,太大了容易过度拟合(overfitting)产生高频噪声。一般情况下我们默认T=2就行。
Mask diameter注意别比颗粒还小就行,不然就会把颗粒信号屏蔽掉,大于box size就是不用mask。
关于二维分类算法,RELION-4.0新版默认使用VDAM algorithm而不是之前的EM algorithm。VDAM算法理论上速度更快,分类效果稍差一些,不支持多MPI运算,酌情选择。我这里因为是很小的数据集,不考虑时间因素,用了传统的EM algorithm,迭代次数Number of EM iterations默认25足够。
Compute选项卡。如果要用ssd加速运算就在Copy particles to scratch directory给一个ssd里的路径,存储服务器一般都是hdd。Use GPU默认是No,如果要使用GPU,可以改成Yes并输入GPU卡号。记得前面说的,MPI数量比GPU数量多一个。
然后点击Run!
2D classification结束后,还是用Subset selection来选择2D类。
注意,我们之前用过Subset selection并且改过Subsets里的参数,RELION会保留这些修改,记得改回来。
我们用第一个Select classes from job并选择2D classification结果的star文件
然后点击Run!
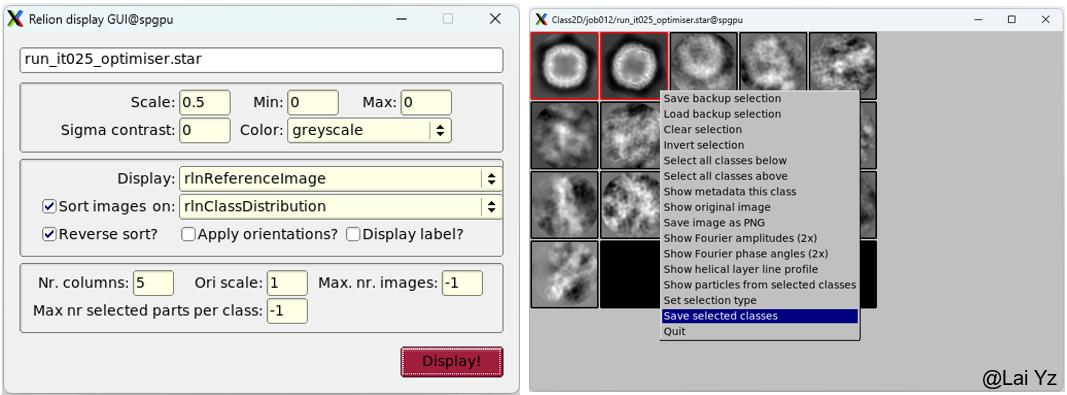
在弹出来的display UI中,Scale参数修改显示的大小,我的屏幕下0.5是比较合适的。勾上Sort images on并选择rlnClassDistribution,会自动按照每一类中颗粒的数量多少排序。勾上Reverse sort从大到小排序。如果使用了Sort排序,那么Max. nr. images就调成-1(无最大数量)。不调也没事,会报一个warning然后忽略就好了。点击Display。
选择质量好的分类,鼠标右键并点击Save selected classes。
实际运算过程中可以进行多轮2D classification,直到2D的结果满意。Tutorial的数据比较简单,一般最多一两轮结果就很不错了。在实际运算中颗粒数量远大于Tutorial的数据,RELION的EM算法进行二维分类速度还是相对比较慢的,可以尝试一下新的VDAM算法,虽然速度比起CryoSPARC还是很慢,而且效果也较差一些。
三维重构
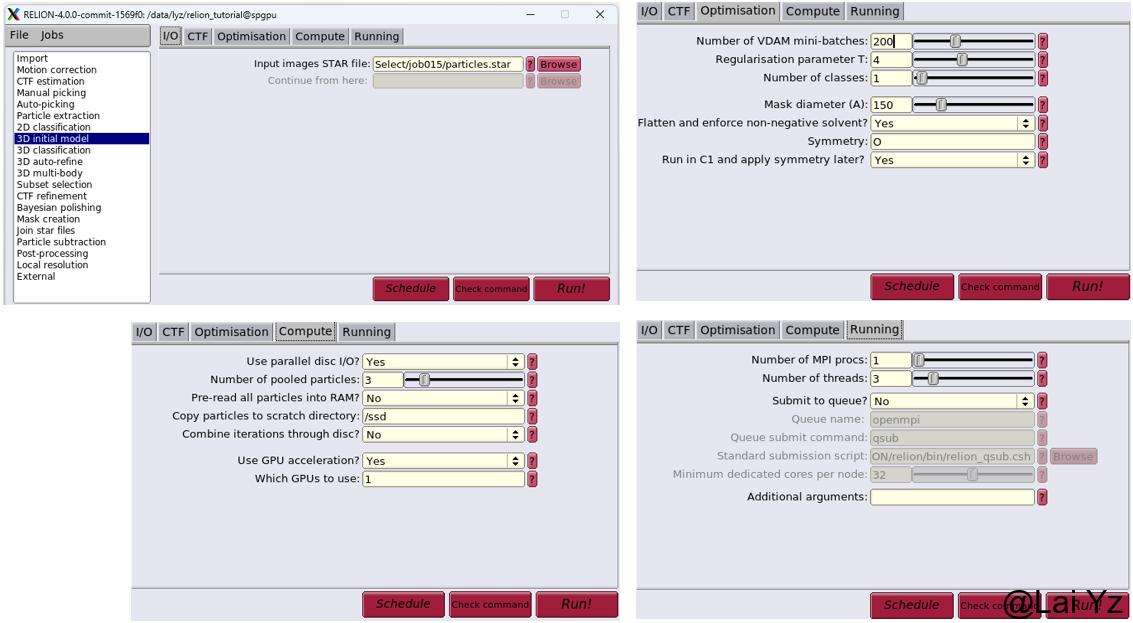
选择3D initio model生成一个初始模型用于后续的3D auto refine。
Input images STAR file选择上一步Subset selection选好的二维类的particles.star。
Optimisation - Symmetry给一个O对称性。
酌情选择ssd、GPU、MPI、threads,然后Run!
运算结果可以通过下载到本地然后用ChimeraX等软件打开。
三维自动细化
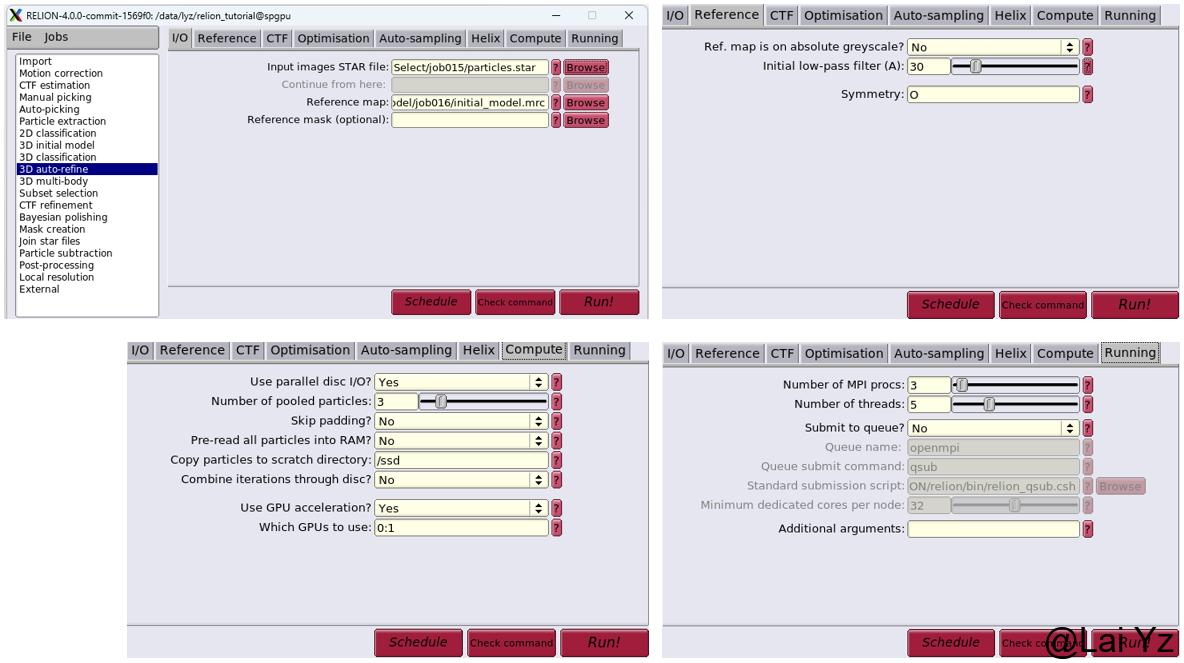
选择3D auto-refine。
Input images STAR file选择上一步Particle extration的particles.star。
Reference map选择刚刚生成的initial model。
Symmetry选择O对称性。
Initial low-pass filter给到30Å就行,会对初始map低通滤波。
Mask diameter记得根据自己的box size和particle diameter调整一下。
Use solvent-flattened FSCs如果选择yes,需要提供一个mask,最后的refine和分辨率会用masked的结果,可以提高分辨率数值。如果这里不做,也可以做完auto-refine了之后再在post-processing里做。
Auto-sampling对于大部分数据来说默认采样角就可以了,但是对于高度对称的颗粒,由于给了对称性,采样角度适当调低一些可能会有好的效果,比如将Initial angular sampling从默认的7.5 degrees调成3.7 degrees,Local searches from auto-sampling默认的1.8 degrees调成0.9 degrees,或许可以获取更好的效果。
GPU选择0和1号。
由于3D auto refine这类的Job都需要用half map的方式评估分辨率和计算,因此至少需要3个MPI procs进行计算,且必需是奇数个(GPU数量+1)。
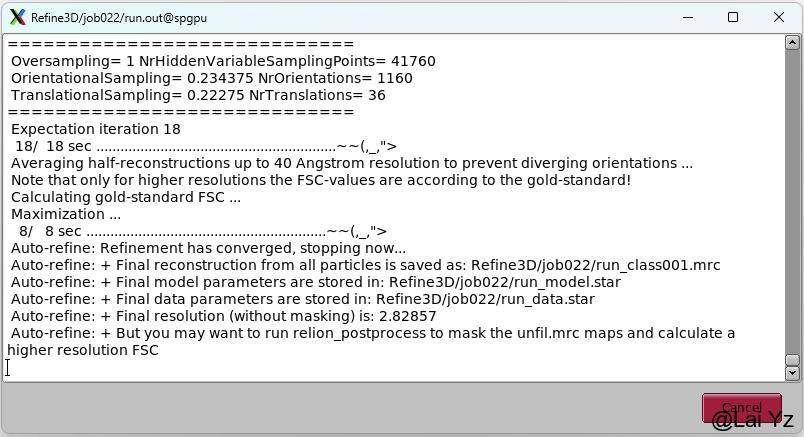
点击Run!。大概需要十几分钟。不出意外的化,这套数据到这里能获得一个2.8Å分辨率的map。
制作Mask以及后处理
刚刚在上一步提到,Use solvent-flattened FSCs这个选项,会最后提供一个masked的结果。如果不提供,也可以在做完auto refine之后再Post-processing。这里我们用Mask creation生成一个mask,然后做Post-processing。
选择Mask creation。
Input 3D map 选择刚刚3D auto-refine的map。
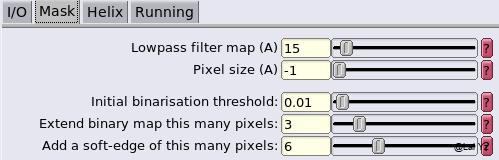
Initial binarisation threshold 把map在Chimera中打开,调节contour level至刚好能看到边缘和一些溶剂信号,这个level就是差不多的Thresholds,一般在0.002-0.02之间。
Extend binary map 会在map外衍生几个pixels以确保覆盖信号,设置为0就会生成一个tight mask。
Add a soft-edge 会柔化mask,因为过于尖锐的边缘信号可能在一些情况下会对FSC校正造成一些影响。
点击Run!。马上就能生成一个Mask。
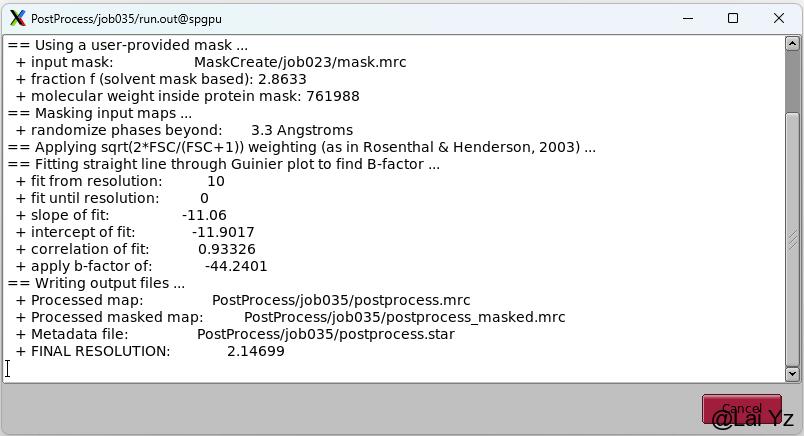
创建好mask后选择Post-processing,将autorefine的map和生成的mask作为Input。sharpen选项卡参数可以默认。
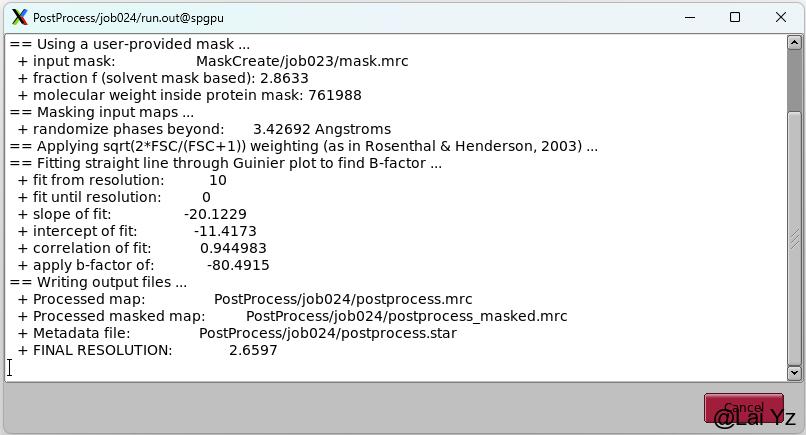
点击Run!。最终获得的分辨率是2.6A。
Post-processing的map(灰色)和没有Post-processing的map(黄色)放在一起比较,可以看到原始map更加smooth,而sharpen过的map明显更加锐利,能更好地看到密度,在搭建model的过程中就能感受到sharpen map的侧链密度能更好地放进去。由于Mask creation和Post-processing这两个job不怎么费时间,所以参数可以调整,Mask的几个参数调整到一个比较满意的效果。
局部CTF细化及贝叶斯抛光
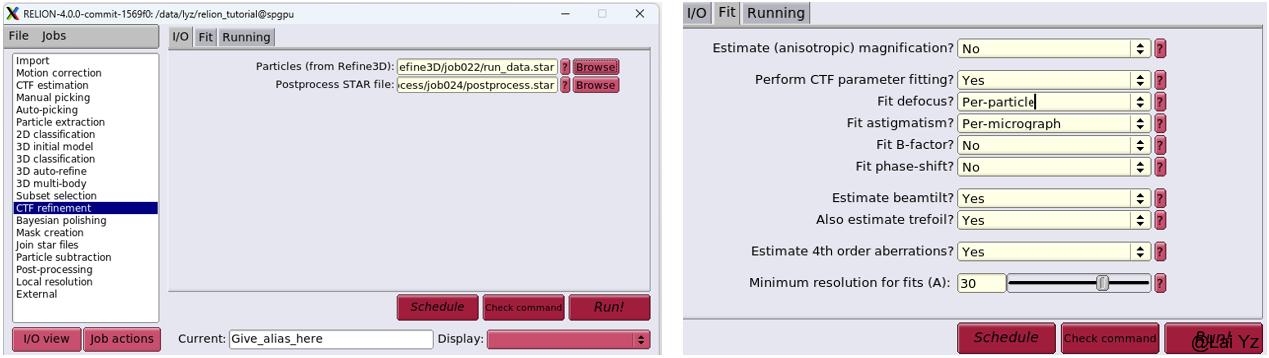
CTF refinement可以估计数据集中的不对称相差(beamtilt和trefoil aberrations)和对称性相差(tetrafoilhe和spherical aberrations),并进一步提高分辨率,但是效果取决于冰层的平整程度(计算per-particle defocus)和显微镜对齐程度(计算aberration)。从3D auto-refine和Post-processing的结果作为Input数据。
Perform CTF parameter fitting选择Yes。
Fit defoucus选择Per-particle。
Fit astigmatism选择Per-micrograph。
Estimate beamtilt和estimate trefoil选择Yes。
点击Run!。
CTF refinement过后再对颗粒进行Bayesian polishing。Bayesian polishing运用Bayesian方法,基于每个颗粒的Beamtilt进行Motion Correction。
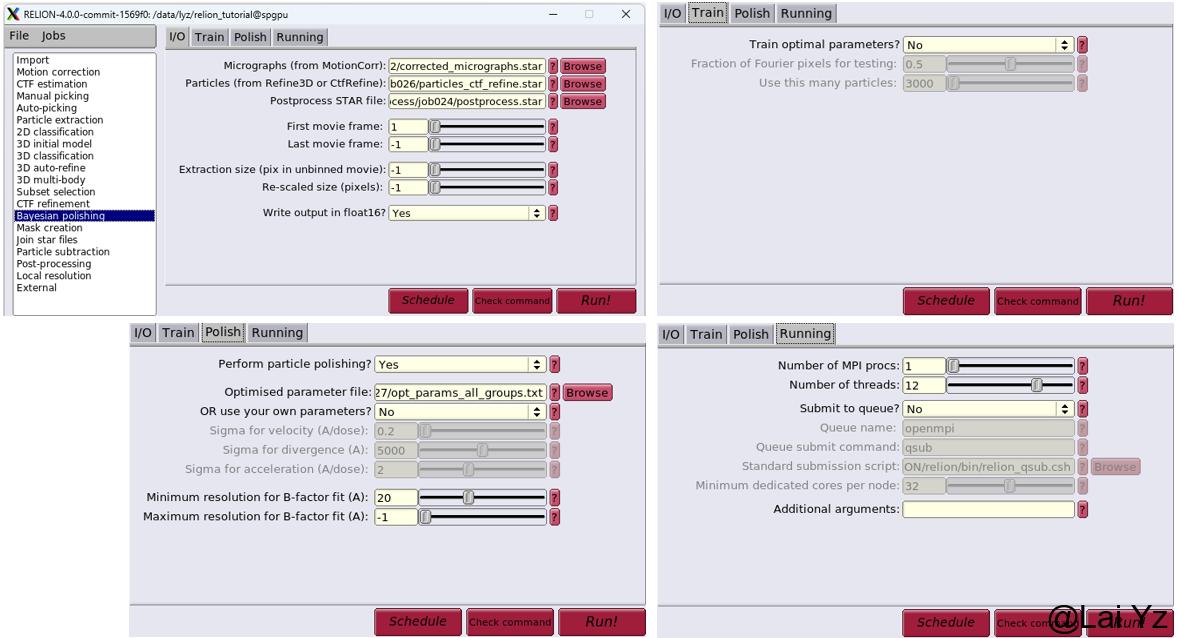
左侧任务选择Bayesian polishing。
I/O的Micrographs Particles STAR file分别输入前面Motion Correction CTF refinement Post-processing的结果。Train的过程比较耗时间,不支持MPI并行运算,需要数小时甚至更久。这里是测试数据,如果不想等也可以仅使用RELION预先训练的通用参数,看看效果。Train选No,Polish选Yes,下面的Sigma用RELION给的通用参数。点击Run!。
如果要是做Train的话Train - Train optimal parameters选择Yes,Polish - Perform particle polishing选项选择No。Run!。然后就会训练一个参数文件出来。
训练完后做Bayesian polishing,区别在于在Polish - Optimised parameter file选择对应的训练出来的参数文件,并且use your own parameters选择No。Run!
Bayesian polishing完成之后再重新跑一遍CTF refinement,然后重新做3D auto-refine和Post-processing等操作。
最终分辨率应该会提高到2.1Å,已经近原子分辨率了。
三维分类
最后来说三维分类,因为这套数据小,而且均一,并不需要三维分类。实际运算中肯定会遇到蛋白有不同的构象,需要用三维分类分开不同Subsets然后分别进行后续的运算。
左侧选择和3D classification。I/O中Input images STAR file选择输入的particle.star,可以是2D classification或者3D auto-refine之类的结果。还需要在Reference map处给一个起始的模型,3D intial model或者3D auto-refine都行,反正在Reference - Initial low-pass filter会起始低通滤波的,默认60Å就行。
Optimisation Sampling Compute里面的参数和2D classification基本都差不多,Regularisation parameter TT值对于三维分类默认是4,有利于分出不同的类,如果想要分出更多差距更小的类,可以增大T值,注意overfitting。
如果你知道自己在做什么,Sampling允许不进行alignment,就如在很多文章中看到的一样。在3D auto-refine已经高精度地找到了角度并且对齐颗粒的情况下,不进行alignment而进行local angular searches来对柔性摆动区域进行分类,适合某一domain刚性而另一头柔性较大的蛋白。
分完类继续后面的refine和后处理部分就行。
从CryoSPARC中导入颗粒集
RELION没有现成的导入cryoSPARC的Job可以用,并且RELION只识别自己的.star文件,而cryoSPARC的文件是.cs后缀格式的。
我们需要用到。
我们服务器上已经装好了,通过以下命令激活:
module load conda 此时用户名前应该显示(base)
conda activate pyem 此时用户名前应该显示(pyem)
这里我们假设cryoSPARC和relion的project分别为/data/lyz/CS-test和/data/lyz/relion_dir。
从cryoSPARC的J20(一个Homogeneous refinement的Job)把颗粒导入relion中计算。还需要的一个cryoSPARC Job是Extract particles,我们假设是J10。
进入cryoSPARC文件夹中J20的目录,用ls命令可以看到两个文件:particle_selected.cs和passthrough_particles_selected.cs。
用csparc2star.py的脚本,把两个文件合并且生成fromcsJ20.star文件(这个文件随便命名,以.star结尾)。然后需要修改一下这个,把文件里的particles.mrc改成particles.mrcs。因为RELION识别mrcs。最后把生成的文件挪到RELION文件夹下。
csparc2star.py particle_selected.cs passthrough_particles_selected.cs fromcsJ20.star
sed -i '/s/particles.mrc/particles.mrcs/g' fromcsJ20.star
mv fromcsJ20.star /data/lyz/relion_dir/
由于有了这个文件,它会在当前目录下去找extract particles micrographs,所以我们要在RELION中新建一个和cryoSPARC里名字一样的J10/extract目录,并把cryoSPARC里对应的extract文件软链接过来,使得软件能搜索到extract particles micrographs。
mkdir -p /data/lyz/relion_dir/J10/extract
cd /data/lyz/relion_dir/J10/extract
ln -s /data//lyz/CS-test/J10/extract/*.mrc .
rename .mrc .mrcs *.mrc
这几步相当于在RELION目录下创建了一个指向原来CryoSPARC中提取的颗粒照片的链接,同时给了一个索引文件star。这样RELION就能靠star指向的地方找到这些颗粒数据。
接下来就可以在RELION里导入.star文件。打开RELION,然后选择Import。
Import raw movies?选择no。
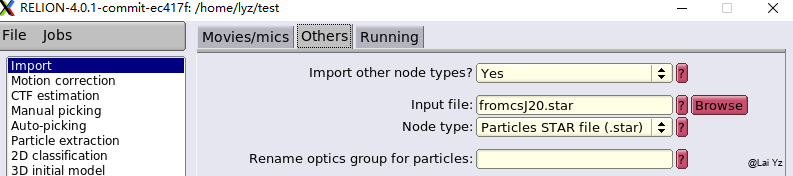
Others选项Import other node types?选择yes。
Input file选择我们生成的star文件。
Node type选择Particles STAR file (.star)。
Run!
这样就成功导入了。可以进行分类和细化任务,但是提取颗粒和贝叶斯抛光这类需要照片和坐标等参数的不行。
还有一种情况是,如果要导入颗粒坐标的话。
csparc2star.py particles_selected.cs J79_passthrough_particles_selected.cs from_csparc_J79.star
sed -i 's/J5\/motioncorrected\/.\{21\}_/MotionCorr\/job002\/Movies\//g' from_csparc_J79.star
sed -i 's/_patch_aligned_doseweighted.mrc/.mrc/g' from_csparc_J79.star
star.py --offset-optics -1 from_csparc_J79.star from_csparc_J79_final.star
用编辑器将rlnOpticsGroupName改为opticsGroup1。
然后提取颗粒的时候选择Or Re-extract particles,将这个star选择。